这个问题涉及马蹄蟹研究的数据。研究中的每只雌性马蹄蟹都有一只雄性螃蟹贴在她的巢穴中。这项研究调查了影响雌蟹是否有其他男性居住在她附近的因素。被认为影响这一点的解释变量包括雌蟹的颜色(C),脊椎状况(S),体重(Wt)和甲壳宽度(W)。
数据文件:crab.txt。
我们将首先拟合仅具有一个自变量:宽度(W)的泊松回归模型

估计的模型是:$ log( hat { mu_i})$ = -3.30476 + 0.16405W ilog(μi^) = - 3.30476 + 0.16405W
估计的β= 0.164的ASE为0.01997,这是小的,并且该斜率在z值为8.216及其低p值的情况下在统计学上是显着的。
如果我们看一下W对Sa的散点图(见下文),我们可能会怀疑一些异常值
您可以考虑其他类型的残差,影响度量(如我们在线性回归中看到的)以及残差图。
以下是运行R代码其他部分的输出的一部分:


从上面的输出中,我们可以看到预测计数(“拟合”)和线性预测变量的值,即预期计数的对数值。
我们也可以看到,尽管预测是有意义的,但模型并不适合。考虑到剩余偏差统计值为567.88和171 df,p值为零,值/ DF = 567.88 / 171 = 3.321远大于1,因此该模型不适合。缺乏适合可能是由于缺少数据,协变量或过度分散。
更改模型
在上述模型中,我们检测到一个潜在的过分散问题,因为比例因子,例如残差偏差的值/ DF远大于1。
回想一下,过度分散的原因之一是异质性,其中每个协变量组合中的主体仍然差异很大。如果是这样的话,是否违背了Poisson回归模型的泊松模型的假设?

上述R程序的输出:
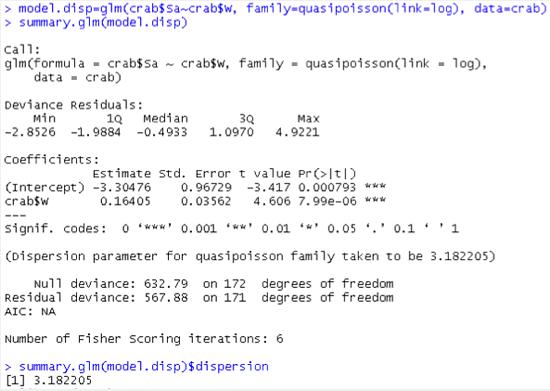
在这个模型中,随机分量在响应具有相同均值和方差的情况下不再具有泊松分布。根据给定的估计值(例如Pearson X 2 = 3.1822),随机分量的变化(响应)大约是平均值的三倍。
除了过度分散之外,如何忽略其他解释变量?我们可以通过添加其他变量来提高拟合度吗?

我们来比较一下这个输出和只有“W”作为预测的模型。我们将“虚拟变量”引入到模型中,以表示具有4级的颜色变量,其中4级作为参考级别。

此外,如果您运行anova(model.disp),从下面的输出中我们可以看到,在考虑宽度后,颜色几乎没有统计上显着的预测因子。
> anova(model.disp) Df Deviance Resid。Df Resid。Dev NULL 172 632.79 W 1 64.913 171 567.88 C1 1 3.130 170 564.75 C2 1 5.400 169 559.35 C3 1 0.004 168 559.34
此模型是否适合数据更好,是否适合过度分散?
R代码的这部分做以下更改:

将此输出的部分与上面的输出相比较,我们将颜色用作分类预测器。我们这样做只是为了记住同一个变量的不同编码会给你不同的拟合和估计值。
现在估计的模型是什么?$ log { hat { mu_i}} $ = -2.520 + 0.1496W - 0.1694C。logμi^ = -2.520 + 0.1496W - 0.1694C。

由于添加协变量没有帮助,过度分散似乎是由于异质性。我们可以用这些数据做些什么吗?
数据分组
我们考虑按宽度分组数据,然后拟合泊松回归模型。这里是按W排序的数据。



数据已分成8个区间,如下面的(分组)数据所示
请注意,“NumCases”是位于特定间隔内的雌性螃蟹的数量,这些雌性螃蟹的宽度由后面限定。“AverWt”是该分组内的平均背宽,“AverSa”是男性卫星总数除以组内的雌蟹总数,“SDSa”和“VarSa”是标准偏差,即“AverSa”的变化。
更改模型
我们还创建了一个变量lcases = log(个案),其中记录了个案数量的对数。这是输出。
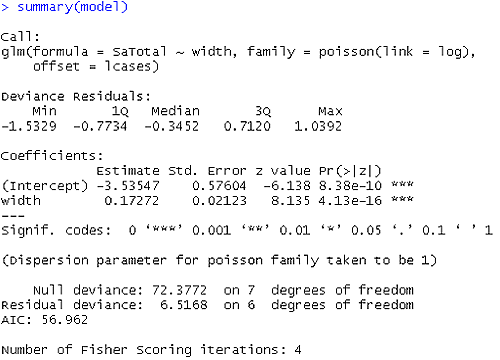
模型现在比以前更好还是更差?它显然更适合。例如,剩余偏差统计值的值/ DF现在是1.0861。
残差分析也显示了良好的拟合度。
我们来比较下图中的观察值和拟合值(预测值):
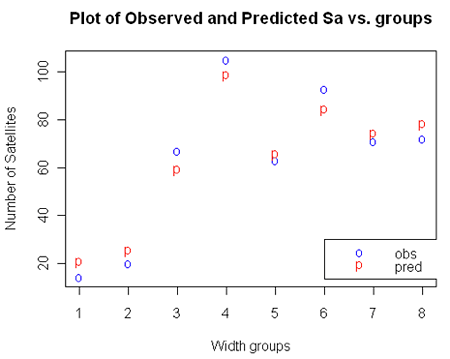
R中的最后两个陈述用于证明我们可以用速率数据的身份链接来拟合泊松回归模型。请注意,该模型不适合分组数据,因为与先前的模型相比,残差偏差统计的值/ DF约为11.649。






















