BP神经网络(Back Propagation Neural Network)Matlab简单实现
- 前言
- 简单了解反向传播(Backwarod Propagation)机制(链式法则)
- 实例分析
- 前向传播(FeedForward Propagation)
- 反向传播(Backward Propagation)/ 误差逆传播
- (一):求解损失/误差相对于每个神经元的梯度
- (二):求解损失/误差相对于每个权值的梯度
- (三):使用梯度下降对权值进行更新
- 动态可视化效果
- 代码实现
前言
前一篇我们介绍了单层感知机,我们知道,它只能解决线性可分数据。对于下图的数据(左为两个同心圆构成的数据,右为异或问题),它将一直迭代且无法收敛。

对于以上两类数据,我们可以通过特征映射,将数据从低维空间映射到高维空间中(例如核函数),使得低维线性不可分问题转化为高维线性可分问题,从而将正负样本分开。如下图(图2来自西瓜书):

我们也可以通过BP神经网络解决这一问题。为了方便大家理解,我们在下文一步一步展开。
简单了解反向传播(Backwarod Propagation)机制(链式法则)
高中时期,我们就学习过关于导数的链式法则(Chain Rule)
而且假设我们已经看过前一篇单层感知机(Single Layer Perceptron)原理及Matlab实现。那么我们可以用一个单层感知机来解释反向传播机制,如下图:

如果输入 经过加权求和
再经过一个激活函数
得到输出的预测值
在这里我们定义的误差损失为真实值和预测值的平方损失:
则我们需要求得损失 关于权值
的梯度,然后通过梯度下降策略,来减小损失。
那么
于是
这样,我们就可以使用梯度下降对感知机的权重进行调整
到达这一步,我们就求出了更新损失的策略,从上面步骤可以看出,误差/损失是沿着神经元一步步逆向回流到每个权值上,于是叫做误差逆传播。
接下来补两张图片直观理解误差反向传播机制(图片来源图解深度学习):


实例分析
如果我们的样本输入为四个二维空间的样本集合:所属的类标签
我们知道单层感知机无法解决上述经典异或问题,接下来我们叠加多层网络,如下图:

其中
- ◍为输入层,
- ◍为隐藏层,为输入层的加权和,
。◍为经过激活函数的输出,h =
。这里的激活函数
为非线性函数。
- ◍为输出层,我们的类别为{0, 1},所以这里面我们使用两个(=类别数)神经元作为预测。这一层我们将激活函数◍也整合进来,即
。
为什么我们的激活函数为非线性的?我们可以想象如果激活函数为线性函数时,那么无论经过多少层,神经网络也只是将输入线性组合后再输出:这就等效于一个线性函数来代替,到头来变成了线性回归模型,隐藏层的作用也就消失了。
而对于非线性的激活函数的作用,可以看作:
- 通过激活函数的非线性映射,将原本处于线性空间数据映射到非线性空间,再经过特征组合,从而形成非线性决策边界,从而逼近复杂函数。
- 通过激活函数,将输出压缩到特定区间内:(0, 1), (-1,1), 等
如何设置神经元数和隐藏层数?
来自Andrew Ng课程:
- 输入层 ◍:神经元数=特征维度。
- 隐藏层 ◍:默认一个。如果选用多个最好保持数目相同,且隐藏层越多,分类效果越好,相应的计算量越大。
- 输出层 ◍:神经元数=类别数。
前向传播(FeedForward Propagation)
下面我们将通过前向传播算法来求解预测值。
首先求解隐藏层的值:。我们可以将偏置(bias)看成值为1,连接权值
的神经元。那么相当于扩充矩阵
:
对于
的权值
,可以写成:
于是隐藏层的输入
可以写成:
经过激活函数(这里先选缺点较多的sigmoid:
),变成
作为隐藏层的输出):
同样,像第一步扩充矩阵,得到
继续前向传播,一般来说输出层节点数与分类的类别数相等。因为我们这里是二分类,所以使用两个节点。这里设置新权值w为:
生成预测标签,这里我们就不展开了:
在这里我们使用二值交叉熵(Cross Entropy Loss)损失函数来作为损失函数:
其中样本数 为数据的真实标签。
所以前向传播可以看成,神经网络从第一层开始,层层向前计算并传播的过程。多层级联的神经网络结合激活函数(非线性的激活函数可以使得决策面不再为直线),使得神经网络具有解决非线性可分数据的能力。
反向传播(Backward Propagation)/ 误差逆传播
(一):求解损失/误差相对于每个神经元的梯度
对于反向传播,我们可以看成将最后损失函数的梯度层层传递给前面层,通过计算图模型(Computational Graph)来将误差层层回溯,关于计算图很多讲解都很明确cs231n、李宏毅老师的ML课程,这里就不说了,主要就是偏微分链式求导法则chain rule性质,下文中也有体现。
已知交叉熵损失函数为:
我们先求交叉熵损失关于输出层中的输入的梯度:
输出层-隐藏层,隐藏层-输入层中的神经元间O - Out, I - In关系可以写为
于是求损失关于神经元的梯度可以写成其中L/∂O已知:
以上述隐藏层的输出◍和输入层◍为例,这里
我们可以观察神经元间的连接线(蓝线、紫线、黑线)来验证为什么这里是点乘和叉乘。
由上步我们很容易得到L◍和L◍的梯度公式。
对于以上梯度的链式求导,我们可以使用雅可比矩阵(Jacobian Matrix)求解:
(二):求解损失/误差相对于每个权值的梯度
我们已经求出来损失函数关于每个神经元的梯度L_◍和L◍,下一步就是求损失函数关于权值的梯度,以便对权值进行更新。
因为权值和下一层神经元的关系可以表示为
所以损失函数对于权值的梯度可以由上一层的神经元传递下来,对上述求导可得:
我们以输入层为例
到此权值的梯度已经获得,接下来就是梯度下降来进行迭代求解。
(三):使用梯度下降对权值进行更新
梯度下降(二)梯度下降(二)
这里就不细说了,本文采用Adam+小批梯度下降进行梯度下降。
动态可视化效果
下面是使用Sigmoid作为激活函数对异或数据分类的结果(1):
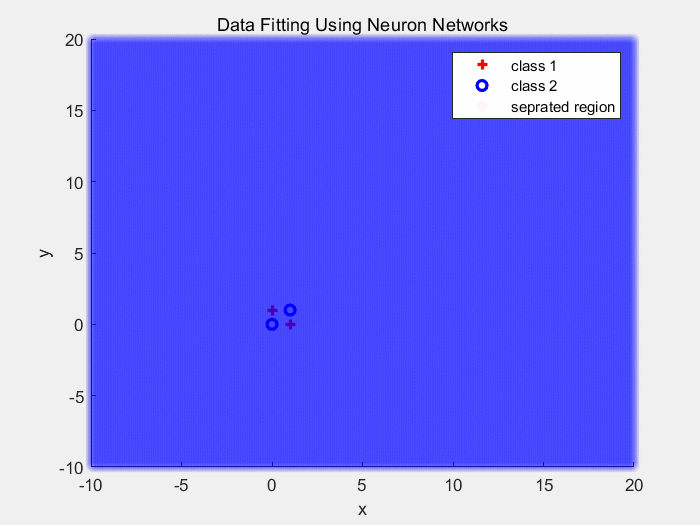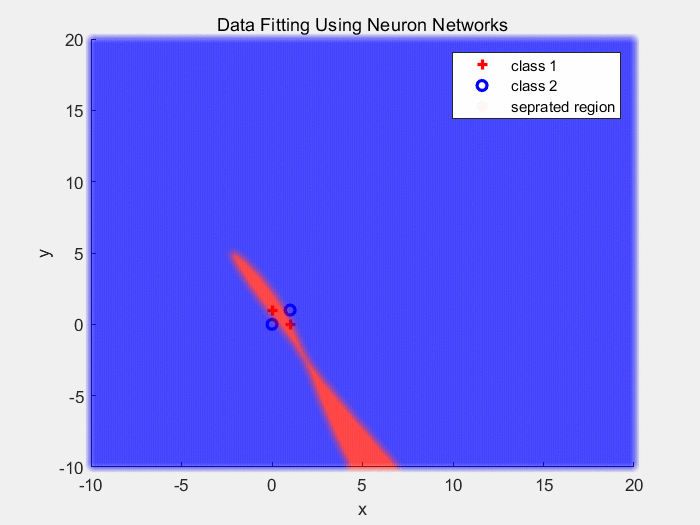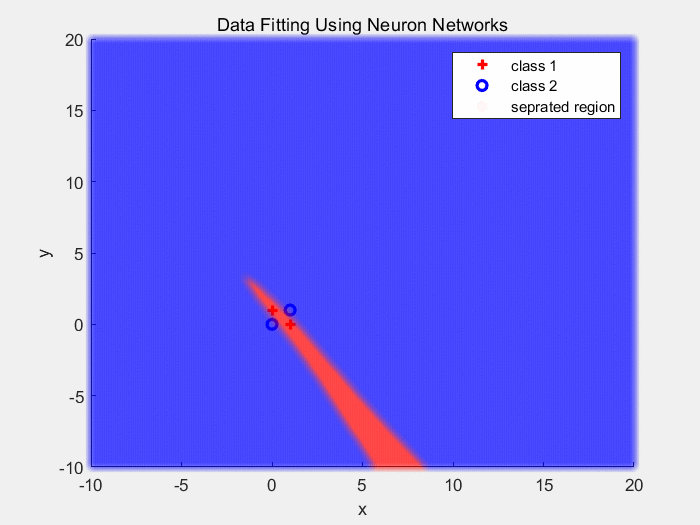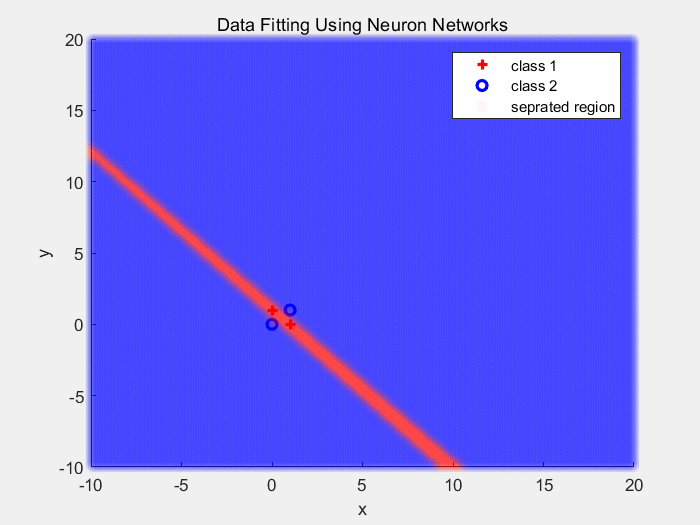
下面是使用Sigmoid作为激活函数对同心圆数据分类的结果(2):
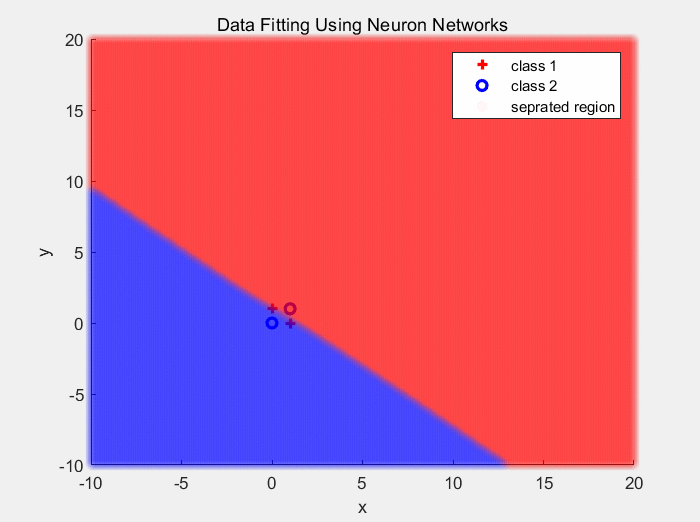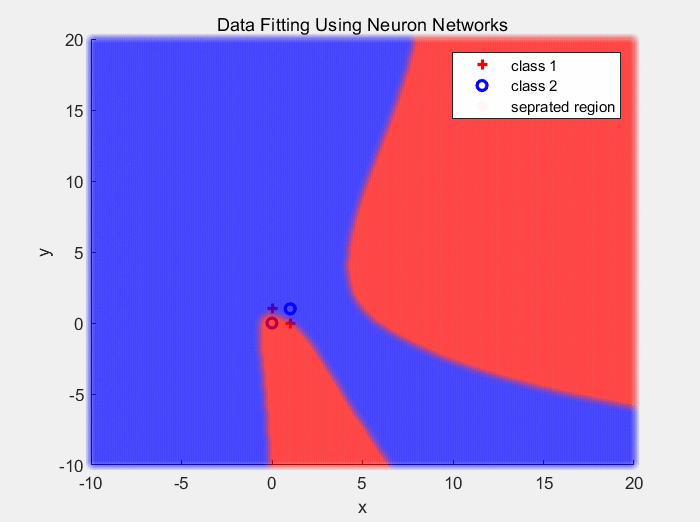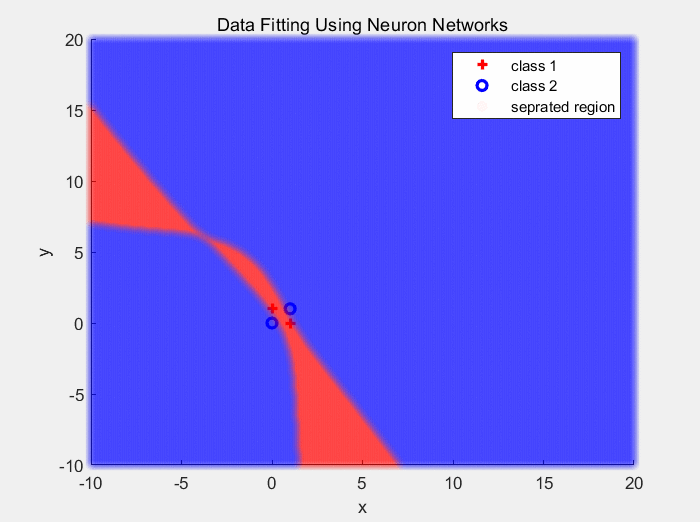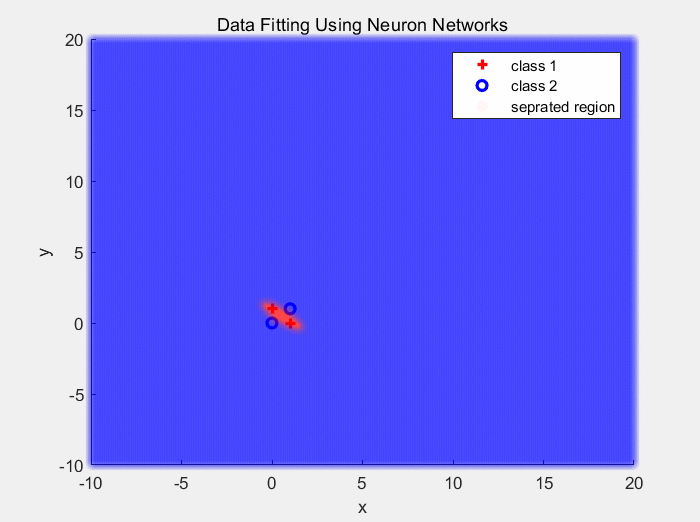
下面是使用ReLU作为激活函数对异或数据分类的结果:

下面是使用Sigmoid作为激活函数对同心圆数据分类的结果:
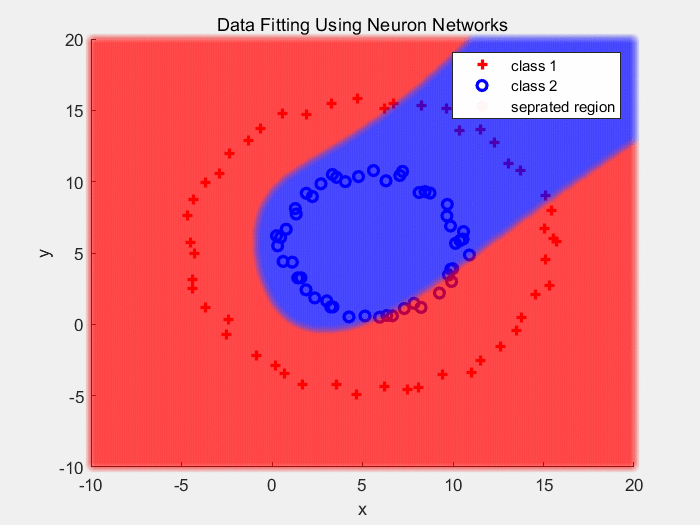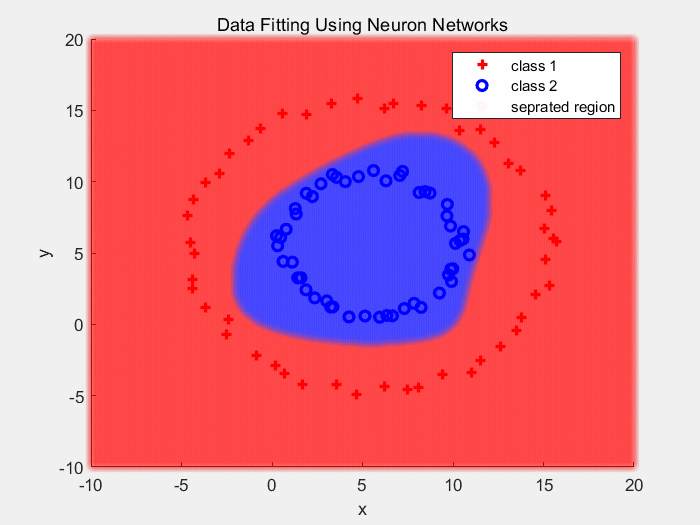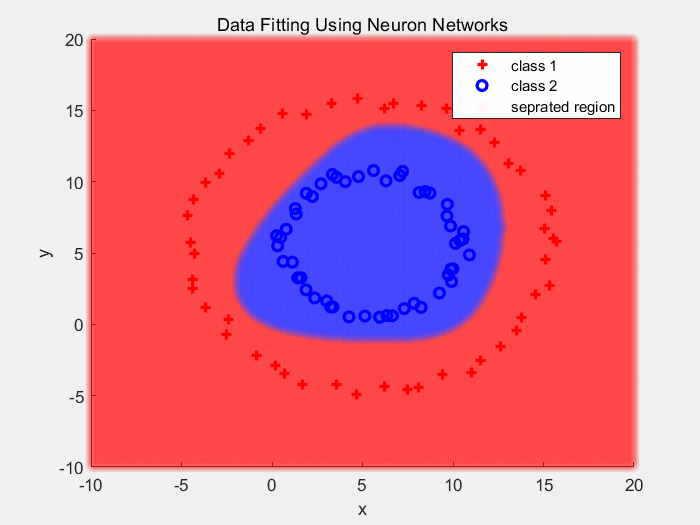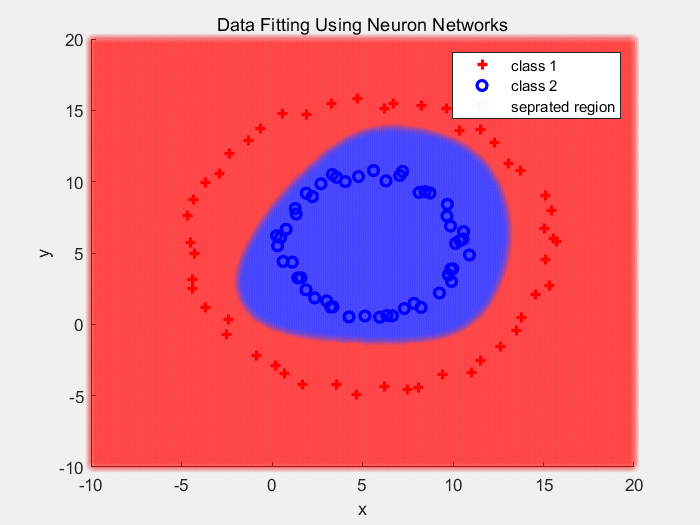
下面是使用ReLU作为激活函数对同心圆数据分类的结果:
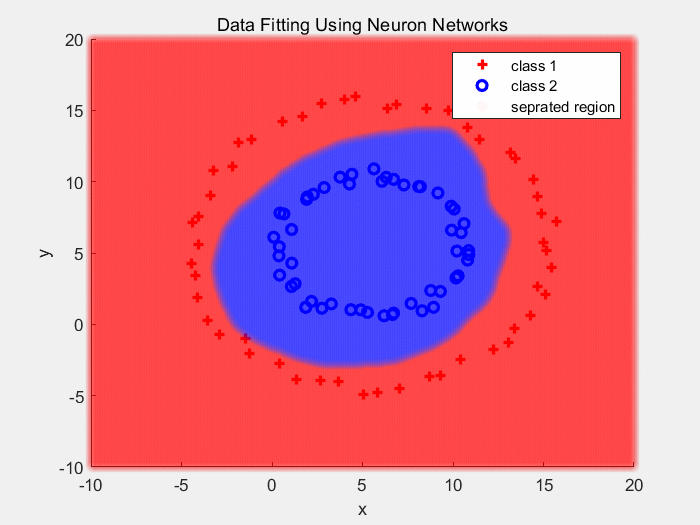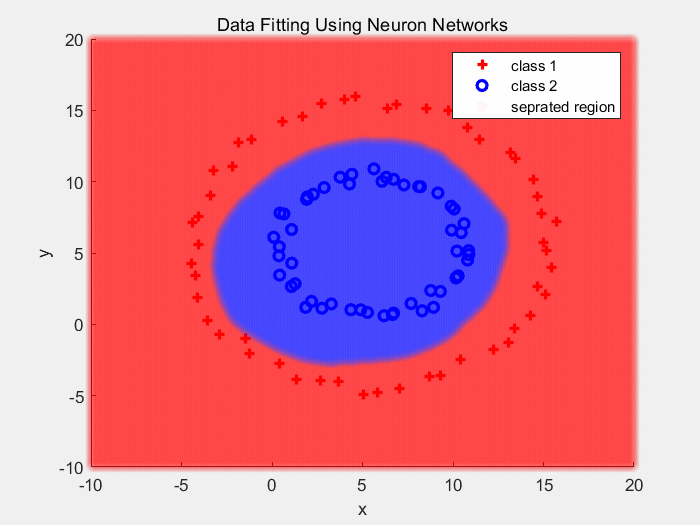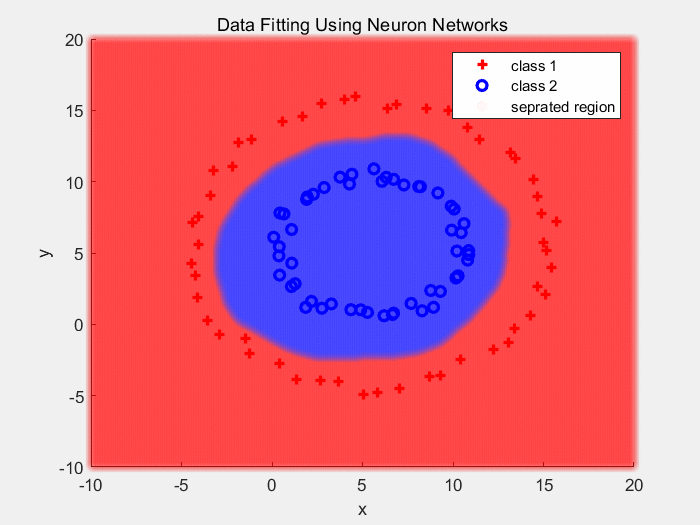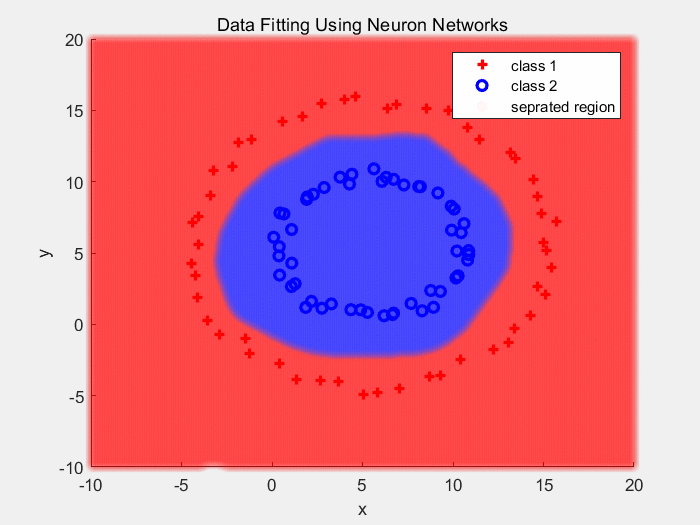
把输入经过多层的传递以及非线性变换后的输出画出来,如下:

可以看到,数据变成线性可分了。
代码实现
本代码参考voidbip ,修改了梯度下降及权值初始化部分,以及增加了ReLU模块,并在其他地方有一定的更改:
% Reference : https://github.com/voidbip/matlab_nn
clc;clear;clf;
X = [0 0 1 1; 0 1 0 1]; % x -> Data set 2-dimension data
flag = [0 1 1 0]; % y -> Flag / label
n = 100; % The first and second data sets
a = linspace(0,2*pi,n/2); % Set the values for x
u = [5*cos(a)+5 10*cos(a)+5]+1*rand(1,n);
v = [5*sin(a)+5 10*sin(a)+5]+1*rand(1,n);
X = [u;v];
flag = [zeros(1,n/2),ones(1,n/2)];
classNum = length(unique(flag)); % How many classes?
[row, col] = size(X); % row -> dimension, col -> size of dataset
NNLayer = [row 20 classNum]; % The structure of our neuron networks
% [1] Initialize weights randomly
w = randInitWeights(NNLayer);
iteration = 10000; % Set our iterations
acMethod = 'SIGMOID'; % Set our activation functions
lambda = 0;
flagMatrix = zeros(classNum,col);
for i = 1 : length(flag)
flagMatrix(flag(i)+1,i) = 1;
end
% - Mini-Batch Gradient Descent Params - %
batchSize = 4;
% - Adam Params - %
eta = 0.002; % Learning rate
s = 0; beta = 0.99; momentum = 0; gamma = 0.9; cnt = 0;
%- draw -%
Range = [-10, 20; -10, 20]; % set the range of dataset
figure(1);
hold on;
posFlag = find(flag == 1);
negFlag = find(flag == 0);
plot(X(1,posFlag), X(2,posFlag), 'r+','linewidth',2);
plot(X(1,negFlag), X(2,negFlag), 'bo','linewidth',2);
[h_region1,h_region2] = drawRegion(Range,w,NNLayer,acMethod);
for i = 1 : iteration
% %i
% cnt = cnt+1;
if(mod(i,100)==0)
delete(h_region1);delete(h_region2);
wFinal = w;
[h_region1,h_region2] = drawRegion(Range,wFinal,NNLayer,acMethod);
title('Data Fitting Using Neuron Networks');
legend('class 1','class 2','seprated region');
xlabel('x');
ylabel('y')
drawnow;
end
% Mini-batch gradient descent + Adam 懒得写成函数了
dataSize = length(X); % obtain the number of data
k = fix(dataSize/batchSize); % obtain the number of batch which has absolutely same size: k = batchNum-1;
batchIdx = randperm(dataSize); % randomly sort for every epoch for achiving sample diversity
flagBatch = flagMatrix(:,batchIdx(1:batchSize));
batchIdx1 = reshape(batchIdx(1:k*batchSize),k,batchSize); % batches which has absolutely same size
batchIdx2 = batchIdx(k*batchSize+1:end); % ramained batch
for batchIdx = 1 : k
valMatrix = ForwardPropagation(X(:,batchIdx1(batchIdx,:)),w,NNLayer,acMethod);
[j,jw] = BackwardPropagation(flagMatrix(:,batchIdx1(batchIdx,:)), valMatrix, w, lambda, NNLayer, acMethod);
cnt = cnt+1;
if j<0.01
break;
end
[sizeW,~] = size(jw);
eps = 10^-8*ones(sizeW,1);
s = beta*s + (1-beta)*jw.*jw; % Update s
momentum = gamma*momentum + (1-gamma).*jw; % Update momentum
momentum_bar = momentum/(1-gamma^cnt);
s_bar = s /(1-beta^cnt);
w = w - eta./sqrt(eps+s_bar).*momentum_bar; % Update parameters(theta)
end
if(~isempty(batchIdx2))
valMatrix = ForwardPropagation(X(:,batchIdx2),w,NNLayer,acMethod);
[j,jw] = BackwardPropagation(flagMatrix(:,batchIdx2), valMatrix, w, lambda, NNLayer, acMethod);
cnt = cnt+1;
%if j<0.01
% break;
%end
[sizeW,~] = size(jw);
eps = 10^-8*ones(sizeW,1);
s = beta*s + (1-beta)*jw.*jw; % Update s
momentum = gamma*momentum + (1-gamma).*jw; % Update momentum
momentum_bar = momentum/(1-gamma^cnt);
s_bar = s /(1-beta^cnt);
w = w - eta./sqrt(eps+s_bar).*momentum_bar; % Update parameters(theta)
end
% Batch gradient descent
% valMatrix = ForwardPropagation(X,w,NNLayer,acMethod);
% [j,jw] = BackwardPropagation(flagMatrix, valMatrix, w, lambda, NNLayer, acMethod);
% w = w-eta*jw;
% j
% if j<0.1
% break;
% end
end
hold off;
%% Initialize Weights Randomly
% input: [2 10 2]
% layer1: 2 neurons + 1 bias.
% layer2: 10 neurons + 1 bias.
% layer3: 2 neurons.
function [w] = randInitWeights(NNLayer)
Len = length(NNLayer); % Obtain the number of layers
shiftLayer = [0 ones(1,Len-1)+NNLayer(1:Len-1)]; % shiftLayer = NNLayer + 1(bias), shiftLayer >> 1。
wCount = NNLayer.*shiftLayer; % The number of weights for previous layer <-> shiftLayer .* NNLayer
w = zeros(sum(wCount),1); % Initialize weight vector
accWIdx = cumsum(wCount); % The index of each layer for weight vector
for i = 2 : Len
eps = sqrt(6)/sqrt(NNLayer(i) + shiftLayer(i));
w(accWIdx(i-1)+1:accWIdx(i)) = eps*(2*rand(wCount(i),1) - 1);
end
end
%% FeedForward Propagation
function [valMatrix] = ForwardPropagation(X, w, NNLayer,acMethod)
[dim, num] = size(X);
Len = length(NNLayer); % Obtain the number of layers
shiftLayer = [0 ones(1,Len-1)+NNLayer(1:Len-1)]; % shiftLayer = NNLayer + 1(bias), shiftLayer >> 1。
accWIdx = NNLayer.*shiftLayer; % The number of weights for previous layer <-> shiftLayer .* NNLayer
ws = cumsum(accWIdx); % The index of each layer for weight vector
accValIdx = [0 cumsum(NNLayer)];
if(dim ~= NNLayer(1))
error("dim of data != dim of input of NN");
end
valMatrix = zeros(sum(NNLayer),num);
valMatrix(1:dim,:) = X;
for i = 2: Len
%curLayerW = reshape(w(ws(i-1)+1:ws(i)),NNLayer(i),shiftLayer(i))';
curLayerW = reshape(w(ws(i-1)+1:ws(i)),shiftLayer(i),NNLayer(i));
valMatrix(accValIdx(i)+1:accValIdx(i+1),:) = activateFunc(curLayerW'*[ones(1,num);valMatrix(accValIdx(i-1)+1:accValIdx(i),:)],acMethod);
end
end
%% Backward Propagation
function [CELoss,jw] = BackwardPropagation(y, valMatrix, w, lambda, NNLayer, acMethod)
Len = length(NNLayer);
[~,num] = size(y);
gradX = zeros(sum(NNLayer(2:end)),num);
jw = zeros(length(w),1);
% CrossEntropy to calculate loss
% Output values: valMatrix(end-NNLayer(end)+1:end,:)
y_hat = valMatrix(end-NNLayer(end)+1:end,:) + 1e-7;
% This is Cross Entropy Loss value
CELoss = -sum(sum(y.*log(y_hat)+(1-y).*log(1-y_hat)))/num;
CELoss = CELoss + ((lambda*sum(w.^2))/(2*num)); % Regularization term
% Easy way for sigmoid function
gradX(end-NNLayer(end)+1:end,:) = y_hat - y; % Obtain the gradient of Cross Entropy / back to y_hat
%gradCE = -(y./y_hat-(1-y)./(1-y_hat));
%gradX(end-NNLayer(end)+1:end,:) = gradCE.*calculateGrad(y_hat,'Sigmoid');
shiftLayer = [0 ones(1,Len-1)+NNLayer(1:Len-1)]; % shiftLayer = NNLayer + 1(bias), shiftLayer >> 1。
accWIdx = NNLayer.*shiftLayer; % The number of weights for previous layer <-> shiftLayer .* NNLayer
ws = cumsum(accWIdx); % The index of each layer for weight vector
gradIdx = [0 cumsum(NNLayer(2:end))]; % Obtain the gradient for each neurons except which in the first layer
ai=[0 cumsum(NNLayer)];
% -- Calculate the gradient of neurons -- %
for i = Len:-1:3
%curLayerW = reshape(w(ws(i-1)+1: ws(i),:),NNLayer(i), shiftLayer(i))'; % Obtain weights between current adjacent layers
curLayerW = reshape(w(ws(i-1)+1: ws(i),:), shiftLayer(i),NNLayer(i)); % Obtain weights between current adjacent layers
curLayerW4X = curLayerW(2:end,:); % Remove the gradients of biases
gradBack = gradX(gradIdx(i-1)+1:gradIdx(i),:); % Get gradients from the next layer
%gradSigmoid = calculateGrad(valMatrix(ai(i-1)+1:ai(i),:),acMethod);
%gradX(gradIdx(i-2)+1:gradIdx(i-1),:) = curLayerW4X*gradBack.*gradSigmoid; % Calculate the gradient of neurons in current layer.
gradActiveFunc = calculateGrad(valMatrix(ai(i)+1:ai(i+1),:),acMethod);
gradX(gradIdx(i-2)+1:gradIdx(i-1),:) = curLayerW4X*(gradActiveFunc.*gradBack); % Calculate the gradient of neurons in current layer.
end
% -- Calculate the gradient for weights -- %
for i = Len:-1:2
temp = zeros(accWIdx(i),num);
for cnt = 1:num
%temp(:,cnt) = kron([1; valMatrix(ai(i-1)+1:ai(i),cnt)],gradX(gradIdx(i-1)+1:gradIdx(i),cnt));
temp(:,cnt) = kron(gradX(gradIdx(i-1)+1:gradIdx(i),cnt),[1; valMatrix(ai(i-1)+1:ai(i),cnt)]);
end
jw(1+ws(i-1):ws(i))= sum(temp,2);
end
jw = jw/num;
jw=jw + lambda*w/num;
end
function val = activateFunc(x,acMethod)
switch acMethod
case {'SIGMOID','sigmoid'}
val = 1.0./(1.0+exp(-x));
case {'TANH','tanh'}
val = tanh(x);
case {'ReLU','relu'}
val=max(0,x);
case {'tansig'}
val=2/(1+exp(-2*x))-1
otherwise
end
end
function val = calculateGrad(x,acMethod)
switch acMethod
case {'SIGMOID','sigmoid'}
val = (1-x).*x;
case {'TANH','tanh'}
error('...'); % TODO...
case {'ReLU','relu'}
val=x>0;
case {'tansig'}
error('...'); % TODO...
otherwise
error('...'); % TODO...
end
end
function [h_region1, h_region2] = drawRegion(Range,w,NNLayer,acMethod)
% Draw region
x_draw=Range(1):0.1:Range(3);
y_draw=Range(2):0.1:Range(4);
[meshX,meshY]=meshgrid(x_draw,y_draw);
[row, col] = size(meshX);
classes = zeros(row,col);
for i = 1:row
valMatrix = ForwardPropagation([meshX(i,:); meshY(i,:)],w,NNLayer,acMethod);
val = valMatrix(end,:)-valMatrix(end-1,:);
classes(i,:) =(val>0)-(val<0); % class(pos) = 1, class(neg) = -1;
end
[row, col] = find(classes == 1);
h_region1 = scatter(x_draw(col),y_draw(row),'MarkerFaceColor','r','MarkerEdgeColor','r');
h_region1.MarkerFaceAlpha = 0.03;
h_region1.MarkerEdgeAlpha = 0.03;
[row, col] = find(classes == -1);
h_region2 = scatter(x_draw(col),y_draw(row),'MarkerFaceColor','b','MarkerEdgeColor','b');
h_region2.MarkerFaceAlpha = 0.03;
h_region2.MarkerEdgeAlpha = 0.03;
end





















