近年来随着基因芯片和DNA微阵列等高通量检测技术的发展,产生了众多的基因表达数据。对这些数据进行有效的分析已经成为后基因组时代的研究重点。一般的聚类是根据数据的全部属性将数据聚类,这种聚类方式称为传统聚类。传统聚类只能寻找全局信息,无法找到局部信息,而大量的生物学信息就隐藏在这些局部信息中。为了更好地在数据矩阵中搜索局部信息,人们提出双聚类概念,目前这种聚类方法得到了越来越广泛的应用。
本文对双聚类提出以来的研究成果进行综述。从基本思想、性能和双聚类结果评价等角度总结重要的双聚类算法类型。
1 双聚类概念自从基因芯片技术产生以来,大量的生物数据需要分析,这些数据大多规格化后以矩阵形式表示和存储。基因芯片数据中隐藏了大量有用的局部模式,为寻找这些信息,CHENG and CHURCH于2000年提出了双聚类(bicluster)概念[1],并给出了双聚类的定义:定义1:设X为基因集,Y为对应的表达条件集。aij为基因表达数据矩阵A中的元素。设I、J分别为X、Y的子集,则(I,J)对指定的子矩阵AIJ具有以下平均平方残基:
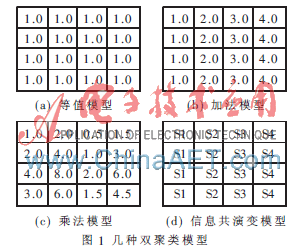
双聚类的目的就是在基因表达数据矩阵中寻找满足条件的子矩阵,使得子矩阵中基因集在对应的条件集上表达波动一致,反之亦然。不同的双聚类算法采用不同的方式度量结果质量,所能找到的双聚类类型是有很大差别的。目前较广泛的模型有四种:矩阵等值模型、矩阵加法模型、矩阵乘法模型和信息共演变模型。图1显示了这几种模型。
2 双聚类算法分类
2.1 基于传统聚类的双聚类
这是一类最基本的双聚类方法,以传统聚类为双聚类的基础,基本思想是通过传统聚类分别对矩阵的行和列进行聚类,然后合并聚类结果。具有代表性的是GETZ G等人[2]提出的耦合双向聚类(Coupled two-way clustering)算法。算法开始于初始矩阵,创建两个集合,一个只包含所有行,另一个只包含所有列。对这两个集合分别运用分层聚类,以产生稳定的行和列的聚类,迭代上述聚类过程来寻找符合条件的稳定子集,将每次产生的稳定基因子集和条件子集分别加在各自的集合中,如此直到没有新的稳定的双聚类出现。基于传统聚类的算法还有很多,如QU[3]等人采用模糊c均值来寻找相似子矩阵模型,通过分别对行和列应用传统聚类得到中间结果,然后合并这些中间结果得到最终双聚类。这类算法实现上较为容易,可以根据不同的需求选择不同的传统聚类算法,算法更加灵活。但这类算法无法完全脱离聚类的全局性,不能很好地寻找局部模式。为克服基于传统聚类算法的缺陷,应该尽量避免传统聚类的全局聚类的思想。如BHATTA A等[4]提出的BCCA算法就很好地避免了全局聚类,算法基于传统聚类的一种距离度量方式,即pearson相关系数,通过计算删除一些使person相关系数明显增加的行列,从而得到双聚类。但BCCA算法不能寻找波动一致而pearson距离较远的双聚类。
2.2 贪心迭代搜索
为摆脱传统聚类的局限性和更好地提高效率,很多算法采用了贪心迭代搜索方法寻找双聚类。CHENG and CHURCH首次使用这种方法寻找双聚类,提出了着名的CC(CHENG and CHURCH)算法[1]。CC算法通过逐步删除可以使子矩阵的平均平方残基降低的行和列,得到一个最初的双聚类,然后逐步添加不会使子矩阵平均平方残基增加的行和列,得到一个较满意的双聚类。为找到更多双聚类,算法使用随机数覆盖已经找到的双聚类,再进行删除和添加过程从而得到指定个数的双聚类结果。算法能够较快地得到用户指定数目的双聚类,但缺陷很明显,随机数替换会改变原始数据,造成结果的不精确,也无法找到重叠的双聚类,而且容易陷入局部最优。YANG[5]等人对CC算法进行了改进,提出了FLOC算法。该算法首先生成一定数量的种子,然后通过计算添加或删除某一行或列,每一步尽量使得双聚类的中间结果增益改变最大。FLOC算法虽然可以找到可重叠的双聚类,但双聚类结果的好坏与运行时间都很大程度地依赖于初始聚类,而这些初始聚类往往都是随机产生的。双聚类的贪心策略效率较高,但聚类结果容易陷入局部最优。为克服贪心策略陷入局部最优的缺陷,一些算法首先采用贪心策略寻找双聚类,然后对找到的双聚类再应用智能优化算法以得到较理想的结果。如STEFAN等人[6]对CC算法进行了改进,即在添加删除过程中好的行列有较大保留概率,反之较小,迭代得到的结果作为种子,应用进化算法优化产生较理想的双聚类。
2.3 双聚类穷举策略
严格地说,采用穷举方式寻找双聚类是不现实的。原数据矩阵的子矩阵数量通常都异常庞大,所以采用穷举策略寻找双聚类算法,多数为穷举小的子矩阵然后合并这些子矩阵的过程。WANG[7]等人提出的δ-Pcluster算法先找到所有基因对和条件对中满足一定条件的双聚类,然后根据条件对的聚类结果对基因对的聚类结果进行剪枝,以基因对条件上的聚类结果剪枝,得到较少的小双聚类构建前缀树,通过后序遍历寻找双聚类。δ-Pcluster算法只为加法模型定义了收敛函数,所以只能限制在加法模型的双聚类上。LIU[8]等人改进了δ-Pcluster算法,采用多个阈值对应多种双聚类模式,可以通过定义多种分组函数,构建了一个OPC树将双聚类的子结果添加入OPC树,通过一次深度优先遍历即可寻找到不同双聚类模式。SAMBA算法[9]是另一个比较重要的基于穷举的双聚类算法,该算法使用统计模型将双聚类问题转化为一个完全平衡二分图搜索问题,再寻找基因表达谱模式,即寻找具有波动一致性的子矩阵问题转化为在二分图中找稠密子图问题。然而,这一算法的重要意义在于:对于基因表达谱进行双聚类分析,实质上是一个NP-hard问题。所以,使用穷举策略的双聚类算法虽然能够找到较优的双聚类,但算法的时间复杂度会随矩阵规模的增大而呈指数增长。因此必须限制双聚类矩阵的大小,同时利用算法技巧优化穷举过程,才能保证算法的效率。
2.4 数学模型方法
利用数学中较成熟的理论或通过建立模型寻找双聚类,一直是研究的热点,也是近年来双聚类发展中的一个趋势。由于双聚类问题的特殊性,即在矩阵中寻找有规律的子矩阵,所以可以较容易地转换成数学模型问题。这类算法中较重要的有LAURA[10]提出的格子模型(Gibbs sampling),它将整个数据集建模为基于聚类表达模式的叠加。也就是说,假如一个突出值属于多个簇,则它等价于这些簇的所有背景值、行影响、列影响的叠加。格子模型更适合确定那些重叠簇,但是这个模型所使用的贪心算法的固有性质却阻碍了这一目标的实现。假设某一值是由多个簇叠加产生的,当确定第一个簇时,实际上这个值受到了所有叠加簇的影响,这意味着这个值将极大地偏离第一个簇的模型。这将导致它被排除到簇外,而实际上它本来是应该在这个簇内的。GU等[11]在Gibbs sampling的基础上提出了贝叶斯双聚类模型(BBC),这种是完全基于模型的一种方法,所以不需要任何阈值参数就能寻找到重叠的双聚类。Kluger[12]等提出的Spectral Biclustering应用线性代数技术寻找数据中的双聚类结构,将在一个条件集上波动一致的基因集看做一种隐藏的棋盘模式,使用特征向量计算寻找这种模式。这类算法的共同之处在于将双聚类问题转化成数学或其他模型,应用各种方法寻找这些模型。数学模型方法寻找双聚类的缺陷也很明显,就是一种数学模型只对应一种或少数的双聚类类型。表1是对以上四种类型优缺点的总结。
2.5 其他双聚类方法
另外的一些较重要方法还有采用分治策略寻找双聚类。其思想是,先将矩阵划分成若干子矩阵,然后对子矩阵进行双聚类,最后合并小的聚类而得到最终结果。这类算法的优点是执行速度较快,但是缺点是算法可能错过一些好的双聚类,因为在发现它们之前,这些双聚类可能己经被分割。模仿生物现象或自然的进化算法越来越普遍,这些方法在数据挖掘和双聚类中有着广泛的应用。如DIVINA等[13]将多目标进化算法应用于双聚类,同时优化多个目标,来发现全局最优解。BRYAN等[14]应用模拟退火模型寻找双聚类,都得到了较好的效果。
3 双聚类结果度量
目前双聚类实验公认的两个数据集分别是:啤酒酵母细胞周期表达值[15]和人类B细胞表达值[16]。双聚类结果质量评价标准有可视化和非可视化标准。双聚类的可视化主要有通过明暗度观察矩阵结构的热图、通过点线连接观察波动性的坐标图、通过基因节点的带有方向性的连接的表达谱图。BARKOW等人[17]开发了一个着名的双聚类算法平台,使用其中的热度图可以较直观地看到数据矩阵的规模,通过明暗度大致了解基因表达的强度。其中也实现了坐标图,这是目前广泛使用的双聚类可视化方式,可直观地看到基因曲线波动的一致性。
非可视化标准往往结合可视化共同度量双聚类算法或双聚类结果的好坏。不同的双聚类策略在时间花费上相差很大,又由于双聚类是NP-hard问题,所以运行时间是度量双聚类算法好坏的一个重要因素。至于双聚类个体的质量,往往会看它是否接近四种基本模型。平均平方残基H是度量结果是否接近模型的较好方式,也是现阶段通常采用的度量手段。双聚类的大小S即包含元素个数也是判断双聚类质量的标准,所以有了许多H的演变形式,例如H/S的形式可有效度量结果,其值越小聚类结果越好。在整个矩阵上找到多个双聚类,所以覆盖矩阵元素的全面性和双聚类结果的重叠性也是重要的质量评价标准。能否找到可重叠的双聚类是设计双聚类算法要考虑的,而结果是否能有效地覆盖矩阵中所有元素也是重要的。另外还有其他的双聚类度量方式,例如在同一双聚类结果上发现了更多属于这个双聚类的基因,而这些基因没有被其他方法发现。
双聚类是个较为年轻的研究领域,近十几年的研究提出了很多有效算法,应用这些算法分析生物芯片数据的过程中也发现了许多有意义的生物学结果。如今双聚类领域虽然主要应用于基因表达数据,但随着研究的发展也将会应用于电子商务等多种领域。由于双聚类问题本身的复杂性,今后依然是个有挑战性的研究课题。
参考文献
[1] CHENG Y, CHURCH G M. Biclustering of expression data[C]. Proc. Eighth Int’l Conf. Intelligent Systems for Molecular Biology (ISMB’00), 2000:93-103.
[2] GETZ G, LEVINE E, DOMANY E. Coupled two-way clustering analysis of gene microarray data[C]. In Proceed ings of the Natural Academy of Sciences USA, 2000:12079-12084.
[3] Qu Jinbin, MICHAEL N, Chen Luonan. Constrained subspace clustering for time series gene expression data[C]. In 4th International Conference on Computational Systems Biology, 2010:9-11.
[4] BHATTACHARYA A, DE R K. Bi-correlation clustering algorithm for determining a set of co-regulated genes[J]. Bioinformatics, 2009, 25(21): 2795-2801.
[5] Yang Jiong, Wang Wei. Enhanced biclustering on gene expression data[C]. In Proceedings of the 3rd IEEE Conference on Bioinformatics and Bioengineering, 2003:321-327.
[6] BLEULER S, PRELIC A, ZITZLER E. An EA framwork for biclustering of gene expression data[C]. Evolutionary Computation, 2004:166-173.
[7] Wang Haixun, Wang Wei, Yang Jiong, et al. Clustering by pattern similarity in large data sets[C]. Proc. The ACM SIGMOD International Conference on Management of Data, 2002:394-405.
[8] Liu Jinze, Wang Wei. Op-cluster: clustering by tendency in high dimensional space[C]. In Proceedings of the 3rd IEEE International Conference on Data Mining, 2003:19-22.
[9] TANAY A, SHARAN R, et al. Revealing modularity and organization in the yeast molecular network by integrated analysis of highly heterogeneous genomewide data[C]. ProcNatl Acad Sic U S A.,2004: 2981-6.
[10] LAZZERONI L, QWEN A. Plaid models for gene expression data[J]. Statistica Sinica, 2002 (12):61-86.
[11] Gu Jiajun, LEE J S. Bayesian biclustering of gene expression data[C]. International Conference on Bioinformatics and Computational Biology, 2007: 25-28.
[12] KLUGER Y, BASRI R, ChANG J T, et al. Spectral biclustering of microarray data:coclustering genes and conditions[J]. Genome Res, 2003,13(4):703-16.
[13] DIVINA F, AGUILAR,RUIZ J S. A multi-objective approach to discover biclusters in microarray data[C]. Proceedings of the 9th annual conference on Genetic and evolutionary computation, 2007: 385-392.
[14] BRYAN K, CUNNINGHAM P, BOLSHAKOVA N. Biclustering of Expression Data Using Simulated Annealing[C].18th IEEE Symposium, 2005: 383-388.
[15] CHO R J, CAMPBELL M J, WINZELER E A, et al. A genome-wide transcriptional analysis of the mitotic cell cycle[J]. Molecular Cell, 1998, 2(1): 65-73.
[16] ALIZADEH A A, Eisen M B, Davis R E, et al. Distinct types of diffuse large B-cell lymphoma identified by gene expression profiling[J]. Nature, 2000(403):503-511.
[17] BARKOW S, BLEULER S, PRELIC A, et al. BicAT:biclustering analysis toolbox[J]. Bioinformatics, 2006,22(10):1282-1283.