决策树与随机森林
二、随机森林
1、Bagging思想
Baggiing是bootstrap aggregating。
- 主要思想:从总体样本中随机选取一部分样本进行训练,通过多次这样的结果,进行投票获取平均值作为结果输出。极大避免不好样本数据,提高准确度。
- bootstrap方法:从样本集进行有放回的重采样。
(案例:假设有1000个样本,如果按照以前的思维,是直接把这1000个样本拿来训练,但现在不一样,先抽取800个样本来进行训练,假如噪声点是这800个样本以外的样本点,就很有效的避开了。重复以上操作,提高模型输出的平均值。)
2、随机森林
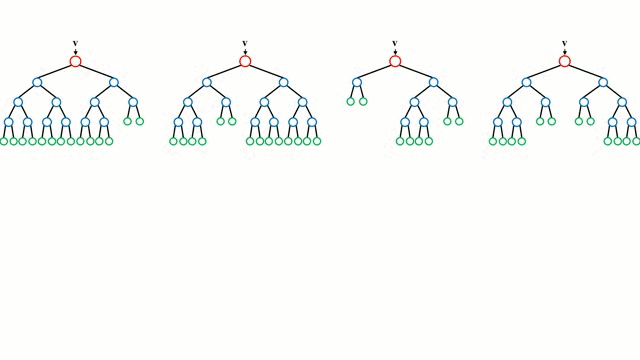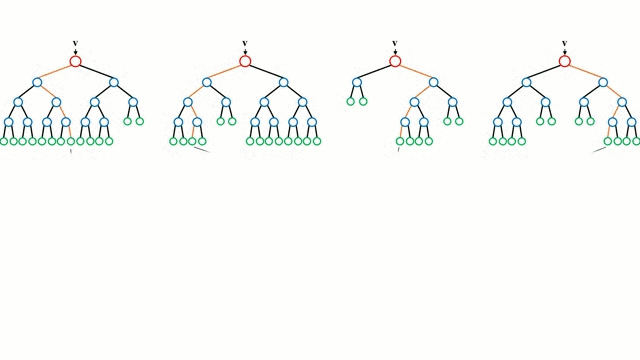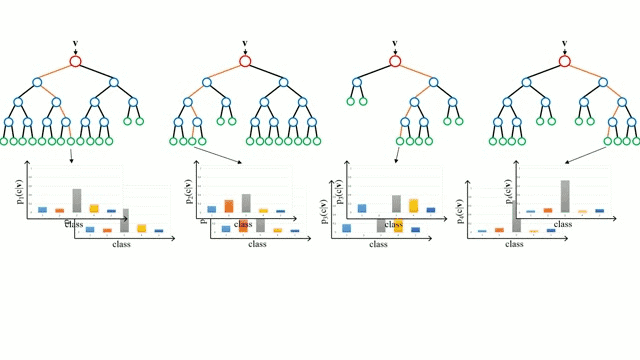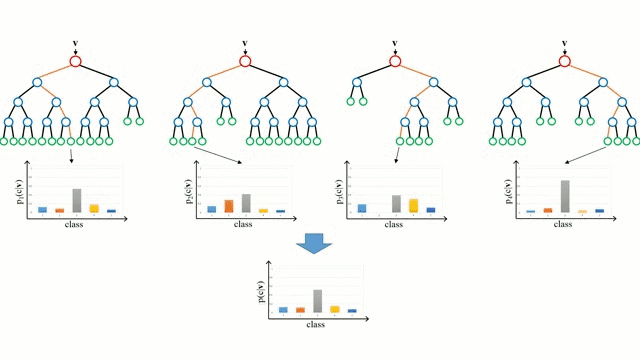
Random Forest(随机森林)是一种基于树模型的Bagging的优化版本,一棵树的生成肯定还是不如多棵树,因此就有了随机森林,解决决策树泛化能力弱的特点。
随机森林步骤:
- 样本的随机:从样本集中用Bootstrap随机选取n个样本
- 特征的随机:从所有属性中随机选取K个属性,选择最佳分割属性作为节点分类器(ID3\C4.5\CART\SVM\LOGISTIC)
- 重复以上两步m次,即建立了m棵分类器(ID3\C4.5\CART\SVM\LOGISTIC)
- 这m个分类器形成随机森林,通过投票表决结果,决定数据属于哪一类(投票机制有一票否决制、少数服从多数、加权多数)
随机选取样本、随机选取特征值、随机选取分类器
3、随机森林分类效果的影响因素
- 森林中任意两个树的相关性:相关性越大,错误率越大;
- 森林中每棵树的分类能力:每棵树的分类能力越强,整个森林的错误率越低。
( 减少特征选择个数m,树的相关性和分类能力会相应的降低; 增大m,两者也会随之增大;)
4、随机森林的优缺点
优点
- 能够处理高纬度的数,并且不用做特征增选择(因为特征集是随机选择的)
- 训练速度快,容易做成并行化方法(训练时树与树之间是相互独立的)
- 在训练过程中,能够检测到特征间的相互影响
- 对于不平和的数据集来书,他可以平衡误差
- 如果有很大一部分的特征遗失,它可以维持准确度
缺点
- 随机森林已经被证明在某些噪音较大的分类或回归问题上过拟合。
- 对于有不同取值的属性的数据,取值划分较多的属性会对随机森林产生更的影响,所以随机随机森林在这种数据上产出的属性权值是不可信的。
5、随机森林中的OOB
(1)OOB:
- oob error(out-of-bag error)袋外错误率是解决随机森林中如何选择最优的m的。
- bagging方法中Bootstrap每次约有1/3的样本不会出现在Bootstrap所采集的样本集合中,当然也就没有参加决策树的建立,把这1/3的数据称为袋外数据oob(out of bag),它可以用于取代测试集误差估计方法。
(2)袋外数据(oob)误差的计算方法
- 假设袋外数据总数为O,用这O个袋外数据作为输入,带进之前生成的随机森林分类器中,分类器会给出O个数据相应的分类。
- 因为这O条数据的类型是已知的,则用正确的分类与随机森林分类器的结果进行比较,统计随机森立分类器分类错误的数目设为X,则袋外数据误差大小=X/O
#随机森林法则
library(randomForest)
library(foreign)
data("iris")
#抽样数据
ind<-sample(2,nrow(iris),replace = TRUE,prob=c(0.7,0.3))
traning<-iris[ind==1,]
testing<-iris[ind==2,]
#训练数据
rf <- randomForest(Species ~ ., data=traning, ntree=100, proximity=TRUE)
#预测
table(predict(rf),traning$Species)
table(predict(rf,testing),testing$Species)
#查看预测的效果
print(rf)
plot(rf)
#查看重要性
importance(rf)
varImpPlot(rf)结果
> table(predict(rf),traning$Species)
setosa versicolor
setosa 32 0
versicolor 0 30
virginica 0 3
virginica
setosa 0
versicolor 4
virginica 30
> table(predict(rf,testing),testing$Species)
setosa versicolor
setosa 18 0
versicolor 0 16
virginica 0 1
virginica
setosa 0
versicolor 2
virginica 14
#查看预测结果
> print(rf)
Call:
randomForest(formula = Species ~ ., data = traning, ntree = 100, proximity = TRUE)
Type of random forest: classification
Number of trees: 100
No. of variables tried at each split: 2
OOB estimate of error rate: 7.07%
Confusion matrix:
setosa versicolor
setosa 32 0
versicolor 0 30
virginica 0 4
virginica
setosa 0
versicolor 3
virginica 30
class.error
setosa 0.00000000
versicolor 0.09090909
virginica 0.11764706
> #查看重要性
> importance(rf)
MeanDecreaseGini
Sepal.Length 7.161310
Sepal.Width 2.252001
Petal.Length 29.192766
Petal.Width 26.700286





















