为什么要用桑基图
桑基图 (Sankey diagram)是一种展现数据之间流动和相互关系的图像,类似于一种网络图,通过不同层级节点之间的连接以及连接边的宽度,展示数据之间的流动关系。

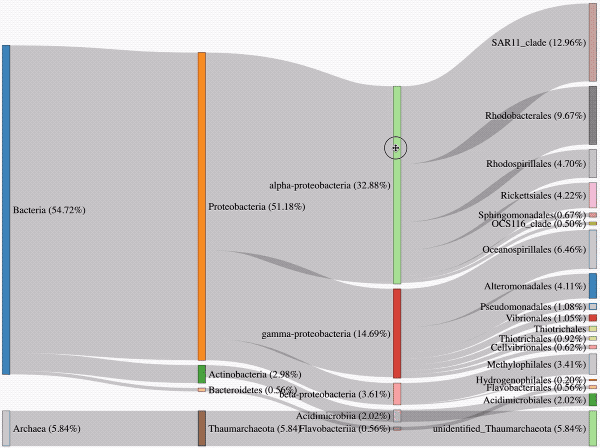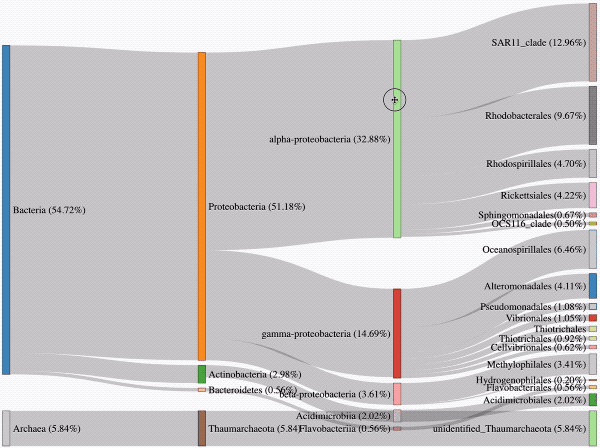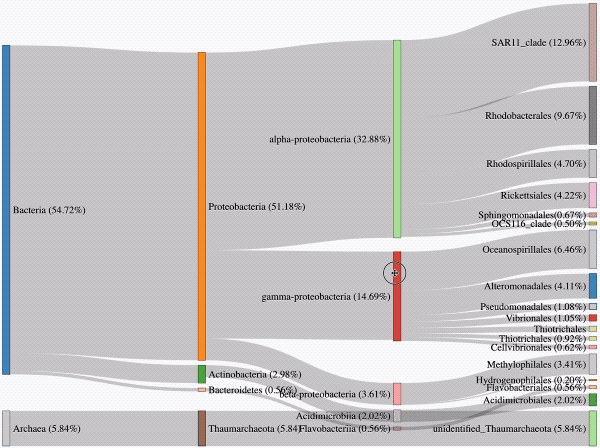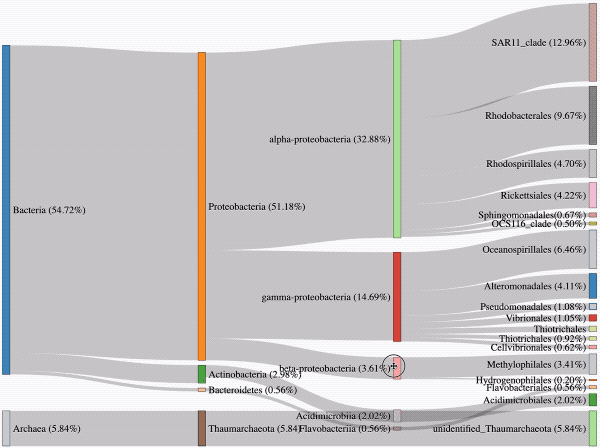
在微生物群落分析中,桑基图可以用来展示不同分类学水平微生物的丰度及其从属关系,可以简单的说是一种包含物种丰度信息的系统发育树。
在一些时间序列或大规模区域样本的研究中,研究人员通常都会非常关注核心微生物组,也就是在一系列时间序列样品或各种不同类型样品中共有的微生物,这些微生物代表了样本中最基本也是最重要的功能微生物。
使用桑基图可以达到同时展示核心微生物组成、系统发育关系以及相对丰度的目的。
桑基图的绘制
可以使用R语言的“d3Network”包进行桑基图的绘制,该包是在R环境下基于D3.j绘制桑基图的专门的包。
还是照例先介绍一下绘图主函数的各项参数。
d3Sankey(Links, Nodes, Source, Target, Value = NULL, NodeID, height = 600, width = 900, fontsize = 7, nodeWidth = 15, nodePadding = 10, parentElement = "body", standAlone = TRUE, file = NULL, iframe = FALSE, d3Script = "http://d3js.org/d3.v3.min.js")各参数意义:
- Links,用于绘图的数据框,给出了节点及其关联信息;
- Nodes,包含节点ID及相关信息的数据框;
- Source,Links文件中指定Source源的向量;
- Target,Links文件中指定Target的向量;
- Value,Links文件中指定节点关联边宽度的向量;
- NodeID,Nodes文件中指定节点ID名称的向量;
- height,输出图像的高度;
- width,输出图像的宽度;
- fontsize,图像中节点标签文字的字号;
- nodeWidth,用于指定每一个节点的宽度;
- nodePadding,介绍里的英语没看明白是什么意思;
- file,输出文件的名称,要输出html格式。
3dNetwork包绘制桑基图需要两个文件,一个是提供节点及其关联信息的数据框,另一个是提供节点名称及其性质的数据框。
我们首先要手动制作一个原始的数据文件,结构类似于网络分析的输入文件,包含三列信息,第一列为source,内容为节点的名称,第二列为target,内容为属于该source的target名称,第三列为value,及其对应target的相对丰度数据。

我的示例数据格式如下,数据是一套时间序列微生物群落样本的共有OTU,之后手动计算各物种的丰度及其对应的系统发育层次信息。
3dNetwork输入的文件要求使用数字来定义source和target的现实顺序,再用nodes文件将节点名称于数字匹配,因此要对输入文件进行一定的处理。
sankey "sankey.txt",header = TRUE,sep = "\t")nodes as.character(sankey$source),as.character(sankey$target))),stringsAsFactors = FALSE)nodes$ID 0:(nrow(nodes)-1)sankey by.x = "source",by.y = "name")sankey by.x = "target",by.y = "name")colnames(sankey) "X","Y","value","source","target")sankey select = c("source","target","value"))nodes select = c("name"))
sankey "sankey.txt",header = TRUE,sep = "\t")
nodes as.character(sankey$source),as.character(sankey$target))),stringsAsFactors = FALSE)
nodes$ID 0:(nrow(nodes)-1)
sankey by.x = "source",by.y = "name")
sankey by.x = "target",by.y = "name")
colnames(sankey) "X","Y","value","source","target")
sankey select = c("source","target","value"))
nodes select = c("name"))
之后就可以绘制图像了。
d3Sankey(Links = sankey,Nodes = nodes,fontsize = 20, Source = "source",Target = "target",Value = "value", NodeID = "name",file = "Sankey.html", width = 1200,height = 900)
d3Sankey(Links = sankey,Nodes = nodes,fontsize = 20,
Source = "source",Target = "target",Value = "value",
NodeID = "name",file = "Sankey.html",
width = 1200,height = 900)
打开输出的html文件,我们会发现部分图像的元素存在一定的重叠,并且有些节点的顺序可能需要修改。

这一步就非常简单了,输出的结果是可交互的,直接鼠标拖动节点模块即可改变其位置。